你是否遇過這樣的情境?使用者憑記憶輸入了關鍵字,例如 `”NIC MACK Test”`(多打了一個 K),或者只輸入了簡短的 `”NIC”`,但原本死板的程式碼卻回傳 `File not found`。
作為開發者,我們如何用 Python 內建的工具,寫出一個既能**容忍錯字**(Fuzzy Match),又能**精準命中**(Exact Match)的智慧搜尋引擎?
這篇文章將帶你經歷一場從「分數低落」到「精準滿分」的演算法優化之旅。
—
## 一、 問題場景:為什麼 `difflib` 給我不及格?
Python 的 `difflib` 是一個強大的序列比對函式庫,但它的評分邏輯(Ratio)有時會違反我們的直覺,特別是在處理**包含關係**的時候。
### 殘酷的現實
讓我們看一個經典案例:使用者想找含有 `”nic”` 的檔案,目標檔名是 `”nic mac test”`。
# %%
import difflib
query = "nic"
target = "nic mac test"
# 計算相似度分數
# 公式: 2 * 匹配長度 / (A長度 + B長度)
# 2 * 3 / (3 + 12) = 6/15 = 0.4
score = difflib.SequenceMatcher(None, query, target).ratio()
print(f"'{query}' vs '{target}' 分數: {score}")
# 輸出: 'nic' vs 'nic mac test' 分數: 0.4# 分數: 0.4
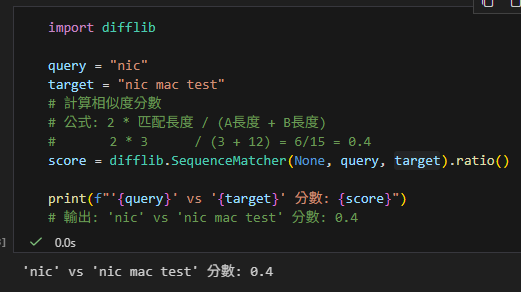
**0.4 分?!**
明明 `”nic”` 100% 完整出現在檔名裡,甚至就是開頭,但因為目標字串太長(稀釋了匹配長度),導致分數不及格。如果不做處理,這個最精確的結果反而會被當成雜訊濾掉。
—
## 二、 解決策略:混合式雙層篩網
為了讓搜尋體驗達到完美,我們不能只依賴單一邏輯。我們需要設計一個**「混合策略」**:
1. **第一層:完全子字串匹配 (Exact Substring Match)**
* 邏輯:只要你包含我,你就是 100 分。
* 解決:「短關鍵字搜尋」分數過低的問題。
* 得分:強制給予 **1.0**。
2. **第二層:模糊相似度匹配 (Fuzzy Match)**
* 邏輯:如果你沒有完全包含,那我們來看看有沒有拼錯字。
* 解決:「手殘打錯字」的問題(如 `MACK` vs `MAC`)。
* 得分:使用 `difflib` 的 **Real Score**(通常 > 0.6)。
—
## 三、 完整程式碼實作
以下是結合了上述邏輯的 Production-Ready 範例代碼。我們還加入了一些資料清洗的技巧(如去除 `.docx` 副檔名)。
import difflib
from pathlib import Path
def smart_search(user_query, file_paths):
results = []
# 預處理:將檔名切割並建立索引
# 使用 p.stem 去除副檔名 (.docx),讓比對更乾淨
file_db = {p.name: p.stem.split("__") for p in file_paths}
print(f"查詢: '{user_query}'\n" + "-"*30)
for filename, segments in file_db.items():
# 轉小寫以忽略大小寫差異
segments_lower = [seg.lower() for seg in segments]
query_lower = user_query.lower()
# --- 策略 A: 完全命中 (給滿分) ---
# 解決 'nic' vs 'nic mac test' 分數只有 0.4 的問題
if any(query_lower in seg for seg in segments_lower):
results.append((1.0, filename, f"【精確命中】包含 '{user_query}'"))
continue # 找到滿分就不用往下算了
# --- 策略 B: 模糊比對 (容忍錯字) ---
# 解決 'NIC MACK' vs 'NIC MAC' 的問題
# cutoff=0.6: 過濾掉像 "common tools" 這種相似度 0.42 的雜訊
matches = difflib.get_close_matches(query_lower, segments_lower,
n=1, cutoff=0.6)
if matches:
best_match = matches[0]
# 這裡才真正使用 difflib 的分數
score = difflib.SequenceMatcher(None, query_lower, best_match).ratio()
results.append((score, filename, f"【模糊建議】修正為 '{best_match}'"))
return results
# --- 測試數據 ---
files = [
Path("C:/Tests/UserA__ProjectX__nic mac test.docx"), # 目標 1 (含有分隔線)
Path("C:/Tests/UserA__ProjectY__switch uart test.docx"), # 干擾項
Path("C:/Tests/Common__Lib__diagnostic tool.docx") # 雜訊 (0.42)
]
# 測試 1: 使用者想找 nic (短關鍵字)
# 結果: 獲得 1.0 分 (原本只有 0.4)
results = smart_search("NIC", files)
results.sort(key=lambda x: x[0], reverse=True)
for score, name, reason in results:
print(f"[{score:.2f}] {name} -> {reason}")
print("\n" + "="*30 + "\n")
# 測試 2: 使用者不小心打錯字 (MACK)
# 結果: 獲得約 0.8 分 (成功救回)
results = smart_search("NIC MACK TEST", files)
results.sort(key=lambda x: x[0], reverse=True)
for score, name, reason in results:
print(f"[{score:.2f}] {name} -> {reason}")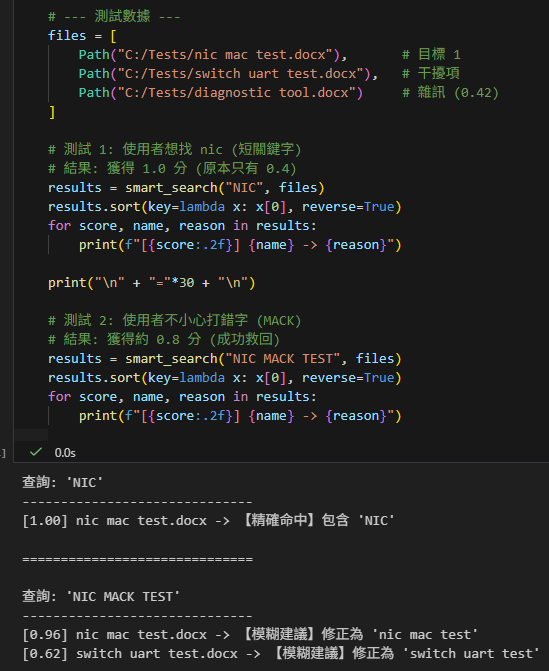
## 四、 成果總結
透過這個簡單的**「混合篩網」**設計,我們達成了一舉兩得的效果:
1. **使用者滿意度**:想精確找的時候(打 `nic`)不會漏掉;打錯字的時候(打 `mack`)也能被糾正。
2. **開發者省力**:不需要引入重量級的 Elasticsearch 或 Lucene,光靠 Python 內建的 `difflib` + `in` 運算符,就能處理 90% 的日常檔名搜尋需求。
這就是**從 0.4 分進化到 1.0 分**的藝術!
—
## 五、 進階知識:difflib.SequenceMatcher 參數全解析
我們在程式中直接使用了 `SequenceMatcher(None, a, b)`,但其實這個類別還有兩個隱藏參數,了解它們能讓你成為真正的專家:
difflib.SequenceMatcher(isjunk=None, a='', b='', autojunk=True)1. **`isjunk` (垃圾過濾器)**
* **預設值**: `None` (不做過濾)。
* **功能**: 允許你定義什麼字元是「垃圾」。
* **應用**: 如果你在比對程式碼,你想忽略「空格」和「TAB」造成的差異,可以設為 `lambda x: x in ” \t”`。這就像是告訴演算法:「這些字不重要,不要因為它們不同就扣分」。
2. **`a`, `b` (比對對象)**
* **類型**: 任何可雜湊 (Hashable) 的序列,通常是字串 (String) 或列表 (List)。
* **功能**: 就是你要比對的兩個主角。
3. **`autojunk` (自動垃圾判斷)**
* **預設值**: `True` (開啟)。
* **功能**: **效能優化機制**。如果某個字元出現頻率過高(例如 C 語言中的 `{` 或 `;`),演算法會自動把它視為垃圾而忽略。
* **注意**: 如果你在比對 **DNA 序列** (只有 A,T,C,G 重複出現),記得把它設為 `False`,否則所有字母都會被當成垃圾,導致比對失敗。
## 六、 歷史小故事:為什麼我們選 0.6?
在我們的程式碼中,我們將 `cutoff` 設為 **0.6**,這其實不是隨便猜的數字。
根據 Python `difflib.SequenceMatcher` 的官方源碼文檔 (Docstring),這個演算法基於 1980 年代的 **”Gestalt Pattern Matching” (格式塔模式匹配)** 理論。它模仿人類的眼睛,先抓出最長的一塊相同區域,而不是死板地逐字比對。
官方文檔明確提到了一條 **Rule of Thumb (經驗法則)**:
> *”As a rule of thumb, a .ratio() value **over 0.6** means the sequences are close matches”*
这解釋了為什麼我們的實驗數據中,雜訊(`diagnostic tool`)的分數會落在 **0.42** (低於 0.6),而真正的拼寫錯誤(`MACK` vs `MAC`)通常都能輕易超過 **0.6**。這個黃金門檻是經過統計驗證的!
推薦hahow線上學習python: https://igrape.net/30afN
difflib.SequenceMatcher?
[31mInit signature:[39m difflib.SequenceMatcher(isjunk=[38;5;28;01mNone[39;00m, a=[33m''[39m, b=[33m''[39m, autojunk=[38;5;28;01mTrue[39;00m)
[31mDocstring:[39m
SequenceMatcher is a flexible class for comparing pairs of sequences of
any type, so long as the sequence elements are hashable. The basic
algorithm predates, and is a little fancier than, an algorithm
published in the late 1980's by Ratcliff and Obershelp under the
hyperbolic name "gestalt pattern matching". The basic idea is to find
the longest contiguous matching subsequence that contains no "junk"
elements (R-O doesn't address junk). The same idea is then applied
recursively to the pieces of the sequences to the left and to the right
of the matching subsequence. This does not yield minimal edit
sequences, but does tend to yield matches that "look right" to people.
SequenceMatcher tries to compute a "human-friendly diff" between two
sequences. Unlike e.g. UNIX(tm) diff, the fundamental notion is the
longest *contiguous* & junk-free matching subsequence. That's what
catches peoples' eyes. The Windows(tm) windiff has another interesting
notion, pairing up elements that appear uniquely in each sequence.
That, and the method here, appear to yield more intuitive difference
reports than does diff. This method appears to be the least vulnerable
to syncing up on blocks of "junk lines", though (like blank lines in
ordinary text files, or maybe "<P>" lines in HTML files). That may be
because this is the only method of the 3 that has a *concept* of
"junk" <wink>.
Example, comparing two strings, and considering blanks to be "junk":
>>> s = SequenceMatcher(lambda x: x == " ",
... "private Thread currentThread;",
... "private volatile Thread currentThread;")
>>>
.ratio() returns a float in [0, 1], measuring the "similarity" of the
sequences. As a rule of thumb, a .ratio() value over 0.6 means the
sequences are close matches:
>>> print(round(s.ratio(), 3))
0.866
>>>
If you're only interested in where the sequences match,
.get_matching_blocks() is handy:
>>> for block in s.get_matching_blocks():
... print("a[%d] and b[%d] match for %d elements" % block)
a[0] and b[0] match for 8 elements
a[8] and b[17] match for 21 elements
a[29] and b[38] match for 0 elements
Note that the last tuple returned by .get_matching_blocks() is always a
dummy, (len(a), len(b), 0), and this is the only case in which the last
tuple element (number of elements matched) is 0.
If you want to know how to change the first sequence into the second,
use .get_opcodes():
>>> for opcode in s.get_opcodes():
... print("%6s a[%d:%d] b[%d:%d]" % opcode)
equal a[0:8] b[0:8]
insert a[8:8] b[8:17]
equal a[8:29] b[17:38]
See the Differ class for a fancy human-friendly file differencer, which
uses SequenceMatcher both to compare sequences of lines, and to compare
sequences of characters within similar (near-matching) lines.
See also function get_close_matches() in this module, which shows how
simple code building on SequenceMatcher can be used to do useful work.
Timing: Basic R-O is cubic time worst case and quadratic time expected
case. SequenceMatcher is quadratic time for the worst case and has
expected-case behavior dependent in a complicated way on how many
elements the sequences have in common; best case time is linear.
[31mInit docstring:[39m
Construct a SequenceMatcher.
Optional arg isjunk is None (the default), or a one-argument
function that takes a sequence element and returns true iff the
element is junk. None is equivalent to passing "lambda x: 0", i.e.
no elements are considered to be junk. For example, pass
lambda x: x in " \t"
if you're comparing lines as sequences of characters, and don't
want to synch up on blanks or hard tabs.
Optional arg a is the first of two sequences to be compared. By
default, an empty string. The elements of a must be hashable. See
also .set_seqs() and .set_seq1().
Optional arg b is the second of two sequences to be compared. By
default, an empty string. The elements of b must be hashable. See
also .set_seqs() and .set_seq2().
Optional arg autojunk should be set to False to disable the
"automatic junk heuristic" that treats popular elements as junk
(see module documentation for more information).
[31mFile:[39m c:\users\appdata\local\programs\python\python311\lib\difflib.py
[31mType:[39m type
[31mSubclasses:[39m推薦hahow線上學習python: https://igrape.net/30afN










![Python, typing: 函數庫規格標註; def addTest(x:float, y:float) -> float: List[資料型態] Set[資料型態] Tuple[資料型態] Dict[str,value的資料型態] Union[資料型態1, 資料型態2] ,函式若有多個輸出值,其實是輸出一個tuple - 儲蓄保險王](https://savingking.com.tw/wp-content/uploads/2022/09/20220907154601_86-520x245.png)
近期留言